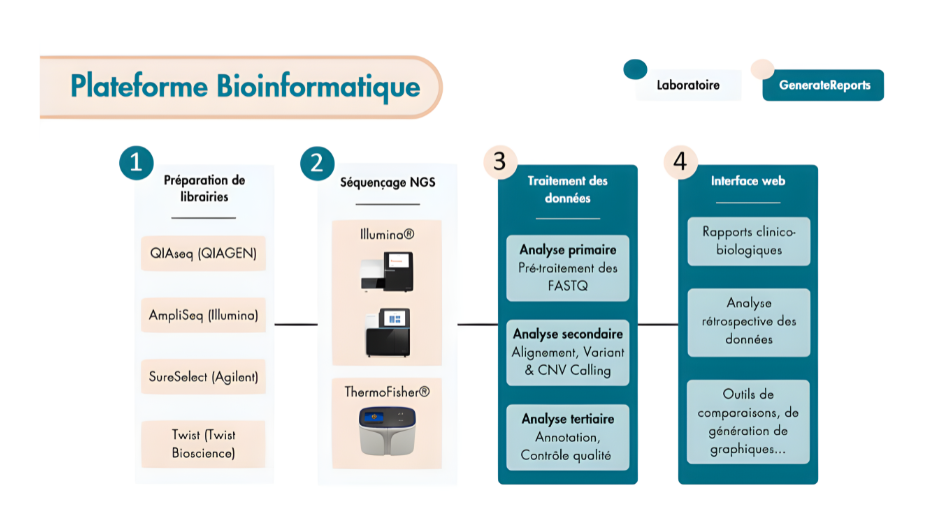
Rapport de Séquençage par Échantillon
Pour chaque échantillon séquencé, un rapport détaillé est généré, offrant une vue complète et transparente des résultats obtenus. Ce rapport intègre les éléments suivants :
- Les données d’identification de l’échantillon
- Un contrôle qualité à l’échelle du Run
- Un contrôle qualité spécifique de l’échantillon
- La liste des variations génétiques détectées et annotées
- Les variations du nombre de copies de gène
- Les informations de traçabilité expérimentale et de traçabilité bio-informatique

Base de Données
Toutes les données sont aussi agrégées dans une base de données permettant la consultation rétrospective des résultats à l’échelle :
- d’un échantillon ou d’un variant
- d’un projet complet
- d’une cohorte de patients
- d’une expérimentation
Des listes de variations peuvent être créées par les biologistes rendant ainsi possible la création de documents, de filtres personnalisés et de certaines analyses automatisées.
La base de données permet aussi le stockage de validations expérimentales externes pour venir confirmer les résultats obtenus par séquençage haut-débit.
Le logiciel autorise l’insertion de données de diverses plateformes de séquençage.
Télécharger la Brochure