Si les nouvelles technologies de séquençage de l’ADN permettent de toujours mieux caractériser les tumeurs, elles génèrent un grand volume de données qu’il faut tenter d’interpréter.
GenerateReports est une plateforme bio-informatique permettant d’analyser des données brutes de séquençage de nouvelle génération dans un rapport clinique synthétisant de manière claire les principaux résultats du séquençage.
Sa conception permet :
- une analyse rétrospective des échantillons biologiques dans le cadre de projets de recherche
- une interprétation des échantillons au quotidien dans le cadre d’une activité diagnostique
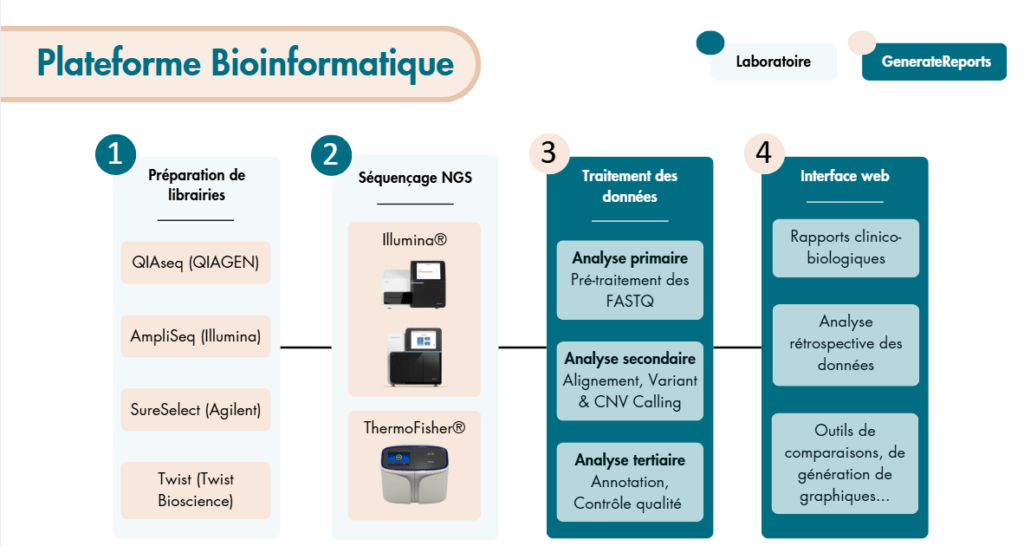
GenerateReports est avant tout un outil modulaire permettant de créer des chaînes de traitement bio-informatique en fonction de la nature des constructions des librairies de séquençage (panels ciblés, exomes, capture, amplicon…) et des questions biologiques posées (diagnostic/recherche, tumor/ctDNA sequencing …).
Il est une aide à l’accréditation au diagnostic de la technologie de séquençage à haut débit (NGS) en permettant un contrôle fin des versions des pipelines bio-informatiques et de leurs dépendances.
GenerateReports intègre la notion de liste de variants afin de permettre des analyses bio-informatiques personnalisées.
Rapport de séquençage pour chaque échantillon
Pour chaque échantillon séquencé, un rapport détaillé est généré, offrant une vue complète et transparente des résultats obtenus. Ce rapport intègre les éléments suivants :
- Les données d’identification de l’échantillon
- Un contrôle qualité à l’échelle du Run
- Un contrôle qualité spécifique de l’échantillon
- La liste des variations génétiques détectées et annotées
- Les variations du nombre de copies de gène
- Les informations de traçabilité expérimentale et de traçabilité bio-informatique.

Toutes les données sont aussi agrégées dans une base de données permettant la consultation rétrospective des résultats à l’échelle :
- d’un échantillon ou d’un variant
- d’un projet complet
- d’une cohorte de patients
- d’une expérimentation
Des listes de variations peuvent être créées par les biologistes rendant ainsi possible la création de documents, de filtres personnalisés et de certaines analyses automatisées.
La base de données permet aussi le stockage de validations expérimentales externes pour venir confirmer les résultats obtenus par séquençage haut-débit.
Le logiciel autorise l’insertion de données de diverses plateformes de séquençage.
| Compatibilité des librairies | QIAseq (QIAGEN) AmpliSeq (Illumina) SureSelect (Agilent) Twist (Twist Biosciences) Pour d’autres librairies, nous contacter |
| Type de données séquencées | Panels de gènes Whole Exome Sequencing (WES) Whole Genome Sequencing (WGS) |
| Variants détectés | Single Nucleotide Polymorphisms (SNPs) Insertion & Deletion (INDEL) Mid-sized INDEL (~100b) Panel/exome/whole genome Copy Number Variant (CNVs) |
| Compatibilité matériel | Séquenceurs Illumina® Séquenceurs ThermoFisher ION S5 |
| Formats des données supportées | Format FASTQ |
| Bases de données utilisées | 1000G ExAC ESP6500siv2 CG46 |
| Scores de prédiction utilisés | ADA_score RF_score Score SIFT Score PolyPhen LRT MutationTaster MutationAssessor FATHMM PROVEAN VEST3 MetaSVM MetaLR MCAP CADD fathmm_MKL_coding GERP Interpro_domain |
Résultats obtenus
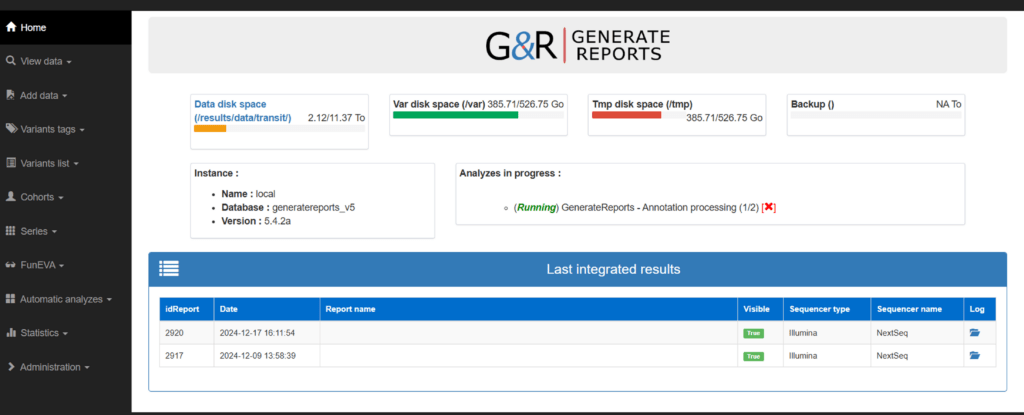
Accéder aux documents GenerateReports
Pourquoi me demande-t-on ces informations ?
En fournissant ces informations, vous acceptez d’être recontacté(e) pour que nous puissions répondre au mieux à vos demandes. ceci nous permet de vous fournir des informations complémentaires personnalisées. Vos données seront supprimées sur simple demande.
Publications liées à GenerateReports
-
UMI-VarCal
PMID: 31985795
-
A recurrent clonally distinct Burkitt lymphoma case
PMID: 30779244
-
Somatic mutations of cell-free circulating DNA
PMID: 30068243
-
Non-invasive monitoring of diffuse large B-cell lymphoma
PMID: 30069017
-
Biological and Clinical Relevance of Associated Genomic Alterations
PMID: 27923841

